Hélice alfa: estructura e importancia funcional
La hélice alfa es la estructura secundaria más sencilla que una proteína puede adoptar en el espacio de acuerdo con la rigidez y libertad de rotación de los enlaces entre sus residuos aminoacídicos.
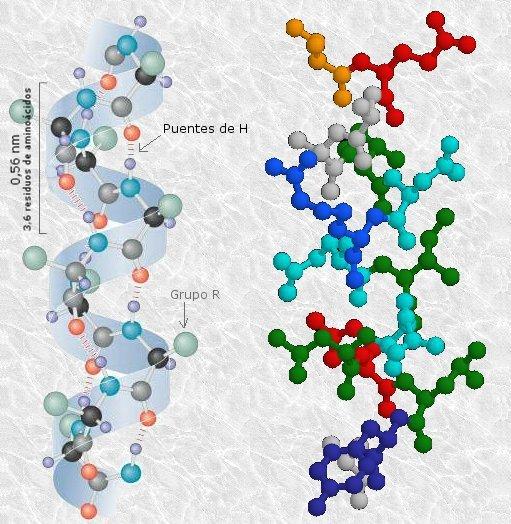
Se caracteriza por la forma espiralada en la que se encuentran dispuestos los aminoácidos, que parecen ordenarse alrededor de un eje longitudinal imaginario con los grupos R hacia el exterior de este.
Las hélices alfa fueron descritas por primera vez en el año 1951 por Pauling y colaboradores, quienes emplearon los datos disponibles sobre las distancias interatómicas, los ángulos de enlace y otros parámetros estructurales de péptidos y aminoácidos para predecir las configuraciones más probables que podrían asumir las cadenas polipeptídicas.
La descripción de la hélice alfa surgió de la búsqueda de todas las estructuras posibles en una cadena peptídica que fuesen estabilizadas por puentes de hidrógeno, donde los residuos fueran estequiométricamente equivalentes y la configuración de cada uno fuese planar, tal y como lo indicaban los datos de resonancia de los enlaces peptídicos que estaban disponibles para la fecha.
Esta estructura secundaria es la más común entre las proteínas, y la misma es adoptada tanto por proteínas solubles como por proteínas integrales de membrana. Se cree que más del 60% de las proteínas existe en forma de hélice alfa o lámina beta.
Índice del artículo
En general, cada giro de una hélice alfa tiene en promedio 3.6 residuos aminoacídicos, lo que equivale más o menos a 5.4 Å de longitud. Sin embargo, los ángulos y las longitudes de giro varían de una proteína a otra con estricta dependencia de la secuencia de aminoácidos de la estructura primaria.
La mayoría de las hélices alfa tienen un giro dextrógiro, pero en la actualidad se sabe que pueden existir proteínas con hélices alfa con giros levógiros. La condición para que ocurra uno u otro es que todos los aminoácidos se encuentren en la misma configuración (L ó D), ya que son estos los responsables de la dirección del giro.
La estabilización de estos importantes motivos estructurales para el mundo proteico está dada por enlaces de hidrógeno. Estos enlaces se dan entre el átomo de hidrógeno unido al nitrógeno electronegativo de un enlace peptídico y el átomo de oxígeno carboxílico electronegativo del aminoácido cuatro posiciones más adelante, en la región N-terminal con respecto a sí mismo.
Cada vuelta de hélice, a su vez, se une a la siguiente por enlaces de hidrógeno, que son fundamentales para conseguir la estabilidad global de la molécula.
No todos los péptidos pueden formar hélices alfa estables. Ello está dado por la capacidad intrínseca de cada aminoácido de la cadena para formar hélices, que se relaciona directamente con la naturaleza química y física de sus grupos R sustituyentes.
Por ejemplo, a determinado pH muchos residuos polares pueden adquirir la misma carga, por lo que no pueden ubicarse consecutivamente en una hélice ya que la repulsión entre ellos implicaría una gran distorsión en la misma.
El tamaño, la forma y la posición de los aminoácidos también son importantes determinantes de la estabilidad helicoidal. Sin ir más lejos, residuos como Asn, Ser, Thr y Cys posicionados en estrecha cercanía dentro de la secuencia también podrían tener un efecto negativo sobre la configuración de la hélice alfa.
De la misma forma, la hidrofobicidad y la hidrofilicidad de los segmentos alfa helicoidales en un péptido determinado dependen exclusivamente de la identidad de los grupos R de los aminoácidos.
En proteínas integrales de membrana abundan hélices alfa con residuos de fuerte carácter hidrofóbico, estrictamente necesarios para la inserción y configuración de los segmentos entre las colas apolares de los fosfolípidos constituyentes.
Las proteínas solubles, por el contrario, poseen hélices alfa ricas en residuos polares, que hacen posible una mejor interacción con el medio acuoso presente en el citoplasma o en los espacios intersticiales.
Los motivos hélice alfa tienen un amplio rango de funciones biológicas. Patrones de interacción específicos entre las hélices juegan un papel crítico en la función, el ensamblaje y la oligomerización tanto de proteínas de membrana como de proteínas solubles.
Estos dominios están presentes en muchos factores de transcripción, importantes desde el punto de vista de la regulación de la expresión genética. También están presentes en proteínas con relevancia estructural y en proteínas membranales que tienen funciones de transporte y/o transmisión de señales de diversa índole.
A continuación, algunos ejemplos clásicos de proteínas con hélices alfa:
La miosina es una ATPasa activada por actina que se encarga de la contracción muscular y de gran variedad de formas de movilidad celular. Tanto las miosinas musculares como las no musculares consisten en dos regiones o “cabezas” globulares unidas entre sí por una larga “cola” alfa helicoidal.
Un tercio del contenido proteico total del cuerpo humano está representado por el colágeno. Es la proteína más abundante del espacio extracelular y tiene como característica distintiva un motivo estructural compuesto por tres hebras paralelas con una configuración helicoidal levógira, que se juntan para formar una hélice triple de sentido dextrógiro.
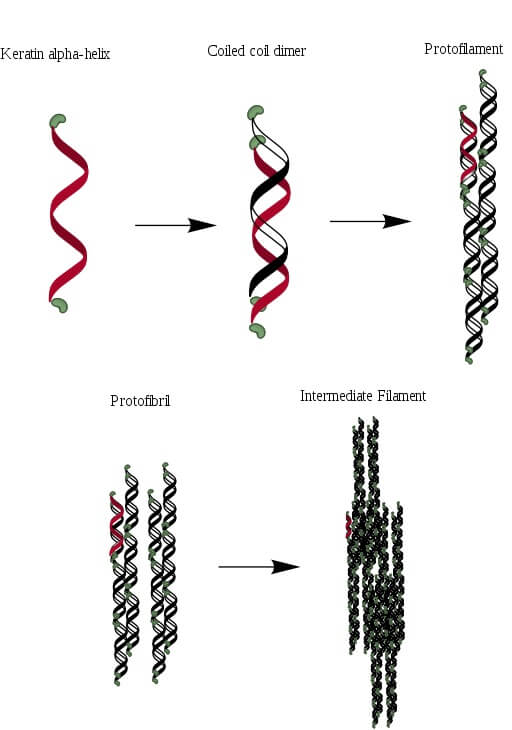
Las queratinas son un grupo de proteínas formadoras de filamentos que son producidas por algunas células epiteliales en los vertebrados. Son el principal componente de las uñas, el cabello, las garras, el caparazón de las tortugas, los cuernos y las plumas. Parte de su estructura fibrilar está formada por segmentos de hélice alfa.
El oxígeno en la sangre es transportado por la hemoglobina. La porción globina de esta proteína tetramérica consiste en dos hélices alfa idénticas de 141 residuos cada una, y de dos cadenas beta de 146 residuos cada una.
Los organismos eucariotas poseen una gran riqueza de proteínas tipo dedos de zinc, que funcionan para diferentes propósitos: reconocimiento de ADN, empaquetado de ARN, activación transcripcional, regulación de la apoptosis, plegamiento de proteínas, etc. Muchas proteínas dedos de zinc poseen hélices alfa como componente principal de su estructura y que son esenciales para su función.
- Aurora, R., Srinivasan, R., & Rose, G. D. (1994). Rules for a-alpha-Helix Termination by Glycine. Science, 264(5162), 1126–1130.
- Blaber, M., Zhang, X., & Matthews, B. (1993). Structural basis of amino acid alpha helix propensity. Science, 260(1), 1637–1640.
- Brennan, R. G., & Matthews, B. W. (1989). The helix-turn-helix DNA binding motif. Journal of Biological Chemistry, 264(4), 1903–1906.
- Eisenberg, D. (2003). The discovery of the structural features of proteins alpha-helix and beta-sheet, the principal. Pnas, 100(20), 11207–11210. Huggins, M. L. (1957). The structure of alpha keratin. Chemistry, 43, 204–209.
- Klement, W., Willens, R., & Duwez, P. (1960). Structure of myoglobin. Nature, 185, 422–427.
- Laity, J. H., Lee, B. M., & Wright, P. E. (2001). Zinc finger proteins: New insights into structural and functional diversity. Current Opinion in Structural Biology, 11(1), 39–46.
- Lodish, H., Berk, A., Kaiser, C. A., Krieger, M., Bretscher, A., Ploegh, H., … Martin, K. (2003). Molecular Cell Biology (5th ed.). Freeman, W. H. & Company.
- Luckey, M. (2008). Membrane structural biology: with biochemical and biophysical foundations. Cambridge University Press. Retrieved from www.cambridge.org/9780521856553
- McKay, M. J., Afrose, F., Koeppe, R. E., & Greathouse, D. V. (2018). Helix formation and stability in membranes. Biochimica et Biophysica Acta – Biomembranes, 1860(10), 2108–2117.
- Nelson, D. L., & Cox, M. M. (2009). Lehninger Principios de Bioquímica. Ediciones Omega (5th ed.).
- Pauling, L., Corey, R. B., & Branson, H. R. (1951). The structure of proteinas: two hydrogen-bonded helical configurations of the polypeptide chain. Proceedings of the National Academy of Sciences of the United States of America, 37, 205–211.
- Perutz, M. F. (1978). Hemoglobin structure and respiratory transport. Scientific American, 239(6), 92–125.
- Scholtz, J. M., & Baldwin, R. L. (1992). The Mechanism of alpha-Helix Formation by Peptides. Annual Review of Biophysics and Biomolecular Structure, 21(1), 95–118.
- Shoulders, M. D., & Raines, R. T. (2009). Collagen Structure and Stability. Annual Review of Biochemistry, 78(1), 929–958.
- Subramaniams, A., Jones, W. K., Gulick, J., & Neumannli, J. (1991). Tissue-specific Regulation of the alpha-Myosin heavy chain gene promoter in transgenic mice. The Journal of Biological Chemistry, 266(36), 24613–24620.
- Wang, B., Yang, W., McKittrick, J., & Meyers, M. A. (2016). Keratin: Structure, mechanical properties, occurrence in biological organisms, and efforts at bioinspiration. Progress in Materials Science. Elsevier Ltd.
- Warrick, H. M., & Spudich, J. a. (1987). Myosin structure and function in cell motility. Annual Review of Cell Biology, 3, 379–421.
- Zhang, S. Q., Kulp, D. W., Schramm, C. A., Mravic, M., Samish, I., & Degrado, W. F. (2015). The membrane- and soluble-protein helix-helix interactome: Similar geometry via different interactions. Structure, 23(3), 527–541
