Superóxido dismutasa: características, estructura, funciones
Las superóxido dismutasas (SOD) o superóxido oxidorreductasas, conforman una familia de enzimas ubicuas en la naturaleza, cuya función principal consiste en la defensa de los organismos aeróbicos frente a radicales libres de oxígeno, especialmente los radicales aniónicos superóxidos.
La reacción que estas enzimas catalizan ocurre virtualmente en todas las células que son capaces de respirar (aeróbicas) y es esencial para la supervivencia de estas, ya que elimina los tóxicos radicales libres del oxígeno, tanto en eucariotas como procariotas.
Muchas enfermedades en los animales se relacionan con la acumulación de diferentes especies reactivas de oxígeno, y lo mismo es cierto para las plantas, dado que el ambiente impone numerosos y constantes tipos de estrés oxidativo que son superados gracias a la actividad de las superóxido dismutasas.
Este grupo de enzimas fue descubierto en 1969 por McCord y Fridovich y desde entonces, avances considerables se han realizado respecto a estas enzimas y las reacciones que catalizan en los seres vivos.
Índice del artículo
Las superóxido dismutasas reaccionan con los radicales superóxido a tasas verdaderamente elevadas, lo que se traduce en una muy efectiva línea de defensa para la remoción de estas moléculas.
En los mamíferos se han descrito al menos tres isoformas para la superóxido dismutasa conocidas como SOD1, SOD2 y SOD3, respectivamente.
Dos de estas isoformas poseen átomos de cobre y de zinc en sus centros catalíticos y difieren entre sí en cuanto a su ubicación: intracelular (citosólico, SOD1 o Cu/Zn-SOD) o con elementos extracelulares (EC-SOD o SOD3).
La isoforma SOD2 o Mn-SOD, a diferencia de las dos anteriores, posee un átomo de manganeso como cofactor y su ubicación parece restringida a las mitocondrias de las células aeróbicas.
Las isoenzimas SOD1 se encuentran principalmente en el citosol, aunque también se han detectado en el compartimiento nuclear y en los lisosomas. Las isoenzimas SOD 3, por el contrario, han sido descritas en el plasma sanguineo de humanos, en la linfa y los fluidos cerebroespinales.
Cada una de estas isoformas está codificada por genes diferentes, pero que pertenecen a la misma familia y su regulación transcripcional está controlada, esencialmente, por las condiciones extra- e intracelulares, que disparan diferentes cascadas de señalización internas.
Las superóxido dismutasas con sitios catalíticos que poseen iones de cobre y zinc o de manganeso no son únicas para los mamíferos, estas también están presentes en otros organismos que incluyen plantas y bacterias de distintas clases.
Existe un grupo adicional de superóxido dismutasas, que no se encuentra en los mamíferos, y que son fácilmente reconocibles, puesto que en su sitio activo poseen hierro en lugar de cualquiera de los tres iones anteriormente descritos para las demás clases de superóxido dismutasas.
En E. coli, la superóxido dismutasa que contiene hierro es una enzima periplásmica encargada también de la detección y eliminación de los radicales libres de oxígeno generados durante la respiración. Esta enzima es similar a la encontrada en las mitocondrias de muchos eucariotas.
Las plantas poseen los tres tipos de enzimas: las que contienen cobre y zinc (Cu/Zn-SOD), las que contienen manganeso (Mn-SOD) y las que contienen hierro (Fe-SOD) en su centro activo y en estos organismos ejercen funciones análogas a las de las enzimas no-vegetales.
Los sustratos de las enzimas superóxido dismutasas son los aniones superóxido, que se representan como O2- y que son compuestos intermediarios en el proceso de reducción del oxígeno.
La reacción que catalizan puede ser contemplada en líneas generales como la transformación (dismutación) de los radicales libres para la formación de oxígeno molecular y peróxido de hidrógeno, que son liberados al medio o empleados como sustrato de otras enzimas, respectivamente.
El peróxido de hidrógeno puede ser eliminado posteriormente de las células gracias a la acción de cualquiera de las enzimas glutatión peroxidasa y catalasa, que también ejercen importantes funciones en la protección celular.
Las isoenzimas superóxido dismutasas en los humanos pueden diferir entre sí en cuanto a ciertos aspectos estructurales. Por ejemplo, la isoenzima SOD1 tiene 32 kDa de peso molecular, mientras que la SOD2 y la SOD3 son homotetrámeros de 95 y 135 kDa de peso molecular, respectivamente.
El otro grupo de superóxido dismutasas, las Fe-SOD presentes en plantas y otros organismos distintos a los mamíferos, son enzimas diméricas de subunidades idénticas, es decir son homodímeros.
En algunas plantas, estas Fe-SOD contienen una secuencia señal N-terminal putativa para el transporte hacia el interior de los cloroplastos y otras contienen una secuencia C-terminal tripeptídica de transporte hacia los peroxisomas, por lo que se presume que su distribución subcelular se restringe a ambos compartimentos.
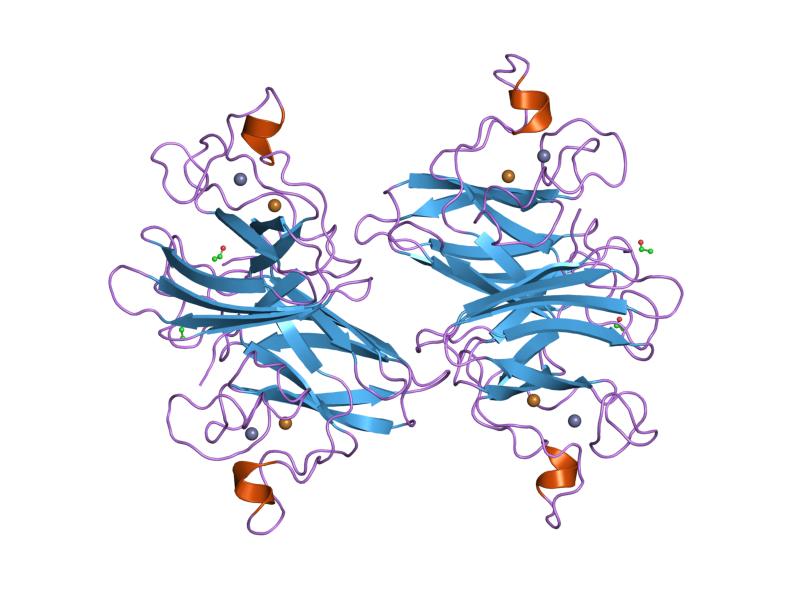
La estructura molecular de los tres tipos de enzimas superóxido dismutasa está conformada, en esencia, por hélices alfa y láminas B-plegadas.
Las superóxido dismutasas defienden a las células, órganos y tejidos corporales de los daños que los radicales libres de oxígeno pueden causar como lo son la peroxidación de lípidos, la desnaturalización de proteínas y la mutagénesis del ADN.
En los animales, estas especies reactivas pueden causar también lesiones cardíacas, acelerar el envejecimiento, y participar en el desarrollo de enfermedades inflamatorias.
Las plantas también requieren de la actividad enzimática esencial de las superóxido dismutasa, puesto que muchas condiciones estresantes del medio ambiente incrementan el estrés oxidativo, es decir, la concentración de especies reactivas deletéreas.
En los humanos y otros mamíferos, las tres isoformas descritas para la superóxido dismutasa tienen diferentes funciones. La isoenzima SOD2, por ejemplo, participa en la diferenciación celular y en la tumorogénesis y también en la protección contra la toxicidad pulmonar inducida por hiperoxia (elevada concentración de oxígeno).
Para algunas especies de bacterias patogénicas, las enzimas SOD funcionan como “factores de virulencia” que les permiten superar muchas barreras de estrés oxidativo a las que pueden enfrentarse durante el proceso de invasión.
Una disminución de la actividad superóxido dismutasa puede darse por varios factores, tanto internos como externos. Algunos se relacionan con defectos genéticos directos en los genes codificantes para enzimas SOD, mientras que otros pueden ser indirectos, relativos a la expresión de moléculas reguladoras.
Gran número de condiciones patológicas de los seres humanos se relaciona con las enzimas SOD, entre ellos la obesidad, la diabetes, el cáncer y otras.
Con respecto al cáncer, se ha determinado que existe gran número de tipos de tumores cancerígenos que poseen bajos niveles de cualquiera de las tres superóxido dismutasas de mamíferos (SOD1, SOD2 y SOD3).
El estrés oxidativo que la actividad superóxido dismutasa previene, también se asocia con otras patologías articulares como la osteoartritis, la artritis reumatoide. Muchas de estas enfermedades tienen que ver con la expresión de factores que inhiben la actividad SOD, como lo es el factor TNF-α.
- Fridovich, I. (1973). Superoxide Dismutases. Annu. Rev. Biochem., 44, 147–159.
- Johnson, F., & Giulivi, C. (2005). Superoxide dismutases and their impact upon human health. Molecular Aspects of Medicine, 26, 340–352.
- Oberley, L. W., & Bueftner, G. R. (1979). Role of Superoxide Dismutase in Cancer: A Review. Cancer Research, 39, 1141–1149.
- Taylor, P., Bowler, C., Camp, W. Van, Montagu, M. Van, Inzé, D., & Asada, K. (2012). Superoxide Dismutase in Plants. Critical Reviews in Plant Sciences, 13(3), 37–41.
- Zelko, I., Mariani, T., & Folz, R. (2002). Superoxide dismutase Multigene Family: A comparison of the CuZn-SOD (SOD1), Mn-SOD (SOD2), and EC-SOD (SOD3) Gene Structures, Evolution, and Expression. Free Radical Biology & Medicine, 33(3), 337–349.
