Enzimas de restricción: funciones, tipos y ejemplos
Las enzimas de restricción son endonucleasas empleadas por ciertas arqueas y bacterias para inhibir o “restringir” la propagación de virus en su interior. Son especialmente comunes en bacterias y forman parte de su sistema de defensa contra ADN foráneo conocido como el sistema de restricción/modificación.
Estas enzimas catalizan el corte de ADN doble banda en lugares específicos, de forma reproducible y sin el uso de energía adicional. La mayor parte requiere de la presencia de cofactores como el magnesio u otros cationes divalentes, aunque algunas también requieren de ATP o S-adenosil metionina.
Las endonucleasas de restricción fueron descubiertas en el año 1978 por Daniel Nathans, Arber Werner y Hamilton Smith, quienes recibieron el premio nobel de medicina por su descubrimiento. Su nombre generalmente deriva del organismo donde son observadas por primera vez.
Tales enzimas son ampliamente utilizadas en el desarrollo de métodos de clonación de ADN y otras estrategias de biología molecular e ingeniería genética. Sus características de reconocimiento de secuencias específicas y capacidad de corte de las secuencias cercanas a los sitios de reconocimiento hacen de ellas poderosas herramientas en la experimentación genética.
Los fragmentos generados por las enzimas de restricción que han actuado sobre una molécula de ADN particular pueden ser empleados para recrear un “mapa” de la molécula original mediante el empleo de la información sobre los sitios donde la enzima cortó el ADN.
Algunas enzimas de restricción pueden tener el mismo sitio de reconocimiento en el ADN, pero no necesariamente lo cortan de la misma forma. Así, existen enzimas que realizan cortes dejando extremos romos y enzimas que cortan dejando extremos cohesivos, que tienen diferentes aplicaciones en la biología molecular.
En la actualidad existen cientos de diferentes enzimas de restricción disponibles comercialmente, ofrecidas por diferentes casas comerciales; estas enzimas funcionan como tijeras moleculares “a la medida” para diferentes propósitos.
Índice del artículo
Las enzimas de restricción cumplen la función opuesta de las polimerasas, ya que estas hidrolizan o rompen el enlace éster dentro del enlace fosfodiéster entre nucleótidos adyacentes en una cadena nucleotídica.
En la biología molecular y la ingeniería genética son herramientas muy utilizadas para la construcción de vectores de expresión y clonamiento, así como para la identificación de secuencias específicas. También son útiles para la construcción de genomas recombinantes y tienen gran potencial biotecnológico.
Los avances recientes en terapia génica hacen uso corriente de las enzimas de restricción para la introducción de genes determinados en vectores que son vehículos para el transporte de tales genes hacia las células vivas, y que probablemente tienen la habilidad de insertarse en el genoma celular para realizar cambios permanentes.
Las enzimas de restricción pueden catalizar el corte de ADN doble banda, aunque algunas son capaces de reconocer secuencias de ADN simple banda e incluso ARN. El corte ocurre tras el reconocimiento de las secuencias.
El mecanismo de acción consiste en la hidrólisis del enlace fosfodiéster entre un grupo fosfato y una desoxirribosa en el esqueleto de cada hebra de ADN. Muchas de las enzimas son capaces de cortar en el mismo sitio que reconocen, mientras que otras cortan entre 5 y 9 pares de bases antes o después del mismo.
Normalmente estas enzimas cortan en el extremo 5’ del grupo fosfato, dando lugar a fragmentos de ADN con un extremo fosforilo 5’ y un extremo hidroxilo 3’ terminal.
Ya que las proteínas no entran en contacto directo con el sitio de reconocimiento en el ADN, estas deben translocarse sucesivas veces hasta conseguir el sitio específico, quizá por medio de mecanismos “deslizantes” sobre la hebra de ADN.
Durante el corte enzimático, el enlace fosfodiéster de cada una de las hebras de ADN es posicionado dentro de uno de los sitios activos de las enzimas de restricción. Cuando la enzima abandona el sitio de reconocimiento y de corte, lo hace a través de asociaciones transitorias no específicas.
Actualmente se conocen cinco tipos de enzimas de restricción. A continuación, una breve descripción de cada uno:
Estas enzimas son grandes proteínas pentaméricas con tres subunidades, una de restricción, una metilación y otra para el reconocimiento de secuencias en el ADN. Estas endonucleasas son proteínas multifuncionales capaces de catalizar reacciones de restricción y modificación, tienen actividad ATPasa y también topoisomerasa de ADN.
Las enzimas de este tipo fueron las primeras endonucleasas en ser descubiertas, se purificaron por primera vez en la década de 1960 y desde entonces se han estudiado con gran profundidad.
Las enzimas tipo I no son muy empleadas como herramienta biotecnológica, puesto que el sitio de corte puede estar a una distancia variable de hasta 1.000 pares de bases respecto al sitio de reconocimiento, lo que las hace poco fiables en cuanto a reproducibilidad experimental.
Son enzimas compuestas por homodímeros o tetrámeros que cortan el ADN en sitios definidos entre 4 y 8 pb de longitud. Estos sitios de corte son típicamente palindrómicos, es decir, reconocen secuencias que son leídas de las misma forma en ambas direcciones.
Muchas de las enzimas de restricción de tipo II en las bacterias cortan el ADN cuando reconocen su carácter de foráneo, ya que no posee las modificaciones típicas que el ADN propio debería tener.
Son estas las enzimas de restricción más simples ya que no requieren de ningún cofactor más que el magnesio (Mg+) para reconocer y cortar las secuencias de ADN.
La precisión de las enzimas de restricción de tipo II en reconocer y cortar secuencias simples en el ADN en posiciones precisas las hace una de las más empleadas e indispensables en la mayoría de las ramas de la biología molecular.
Dentro del grupo de las enzimas de restricción de tipo II se encuentran múltiples subclases clasificadas de acuerdo con ciertas propiedades que son únicas para cada una. La clasificación de estas enzimas se realiza adicionando letras del abecedario, desde la A hasta la Z siguiendo el nombre de la enzima.
Algunas de las subclases más conocidas por su utilidad son:
Subclase IIA
Son dímeros de subunidades diferentes. Reconocen secuencias asimétricas y son empleadas como precursores ideales para la generación de enzimas de corte.
Subclase IIB
Están compuestas por uno más dímeros y cortan el ADN a ambos lados de la secuencia de reconocimiento. Cortan ambas hebras de ADN en un intervalo de pares de bases más adelante del sitio de reconocimiento.
Subclase IIC
Las enzimas de este tipo son polipéptidos con funciones de división y modificación de las hebras de ADN. Estas enzimas cortan ambas hebras de manera asimétrica.
Subclase IIE
Las enzimas de esta subclase son las más empleadas en la ingeniería genética. Poseen un sitio catalítico y generalmente requieren de un efector alostérico. Estas enzimas necesitan interactuar con dos copias de su secuencia de reconocimiento para realizar un corte eficiente. Dentro de esta subclase se encuentran las enzimas EcoRII y EcoRI.
Las endonucleasas de restricción de tipo III están compuestas solo por dos subunidades, una es responsable del reconocimiento y la modificación del ADN, mientras que la otra se encarga del corte de la secuencia.
Estas enzimas requieren dos cofactores para su funcionamiento: el ATP y el magnesio. Las enzimas de restricción de este tipo poseen dos sitios de reconocimiento asimétricos, translocan el ADN de una manera dependiente de ATP y lo cortan entre 20 a 30 pb adyacentes al sitio de reconocimiento.
Las enzimas de tipo IV son fáciles de identificar ya que cortan ADN con marcas de metilación, están conformadas por varias subunidades diferentes que se encargan de reconocer y cortar la secuencia de ADN. Estas enzimas utilizan como cofactores GTP y magnesio divalente.
Los sitios específicos de corte incluyen cadenas nucleotídicas con residuos de citosina metilada o hidroximetilada en una o ambas hebras de los ácidos nucleicos.
Esta clasificación agrupa a las enzimas tipo CRISPER-Cas, que identifican y cortan secuencias específicas de ADN de organismos invasores. Las enzimas Cas utilizan una hebra de ARN guía sintetizado de CRISPER para reconocer y atacar a los organismos invasores.
Las enzimas clasificadas como tipo V son polipéptidos estructurados por enzimas de tipo I, II y II. Pueden cortar secciones del ADN de casi cualquier organismo y con gran rango de longitud. Su flexibilidad y facilidad de empleo hacen a estas enzimas una de las herramientas más empleadas en la ingeniería genética actualmente junto con las enzimas tipo II.
Las enzimas de restricción han sido utilizadas para la detección de polimorfismos de ADN, especialmente en estudios de genética poblacional y estudios evolutivos empleando ADN mitocondrial, con el fin de obtener información acerca de las tasas de sustituciones nucleotídicas.
En la actualidad, los vectores empleados para la transformación de bacterias con diversos propósitos poseen sitios de multiclonaje donde se encuentran sitios de reconocimiento para múltiples enzimas de restricción.
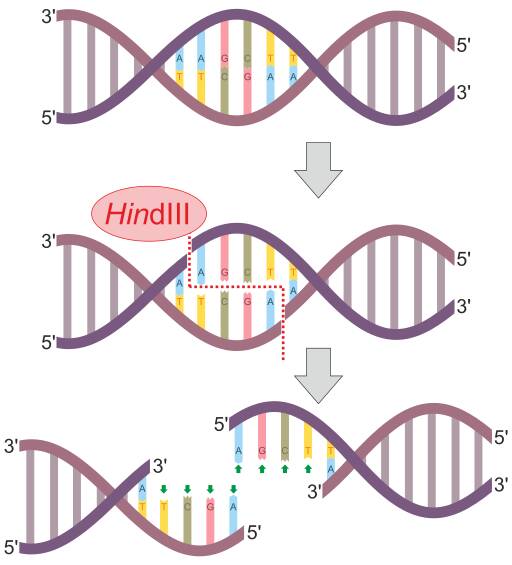
Entre esas enzimas las más populares son EcoRI, II, III, IV y V, obtenidas y descritas por primera vez de E. coli; HindIII, proveniente de H. influenzae y BamHI de B. amyloliquefaciens.
- Bickle, T. A., & Kruger, D. H. (1993). Biology of DNA Restriction. Microbiological Reviews, 57(2), 434–450.
- Boyaval, P., Moineau, S., Romero, D. A., & Horvath, P. (2007). CRISPR Provides Acquired resistance against viruses in prokaryotes. Science, 315(March), 1709–1713.
- Goodsell, D. (2002). The molecular perspective: Restriction Endonucleases. Stem Cells Fundamentals of Cancer Medicine, 20, 190–191.
- Halford, S. E. (2001). Hopping, jumping and looping by restriction enzymes. Biochemical Society Transactions, 29, 363–373.
- Jeltsch, A. (2003). Maintenance of species identity and controlling speciation of bacteria: a new function for restriction/modification systems? Gene, 317, 13–16.
- Krebs, J., Goldstein, E., & Kilpatrick, S. (2018). Lewin’s Genes XII (12 ed.). Burlington, Massachusetts: Jones & Bartlett Learning.
- Li, Y., Pan, S., Zhang, Y., Ren, M., Feng, M., Peng, N., … She, Q. (2015). Harnessing Type I and Type III CRISPR-Cas systems for genome editing. Nucleic Acids Research, 1–12.
- Loenen, W. A. M., Dryden, D. T. F., Raleigh, E. A., & Wilson, G. G. (2013). Type I restriction enzymes and their relatives. Nucleic Acids Research, 1–25.
- Nathans, D., & Smith, H. O. (1975). Restriction Endonucleases in the analysis and restructuring of DNA molecules. Annu. Rev. Biochem., 273–293.
- Nei, M., & Tajima, F. (1981). Dna polymorphism detectable by restriction endonucleases. Genetics, 145–163.
- Pingoud, A., Fuxreiter, M., Pingoud, V., & Wende, W. (2005). Cellular and Molecular Life Sciences Type II restriction endonucleases: structure and mechanism. CMLS Cellular and Molecular Life Sciences, 62, 685–707.
- Roberts, R. (2005). How restriction enzymes became the workhorses of molecular biology. PNAS, 102(17), 5905–5908.
- Roberts, R. J., & Murray, K. (1976). Restriction endonucleases. Critical Reviews in Biochemistry, (November), 123–164.
- Stoddard, B. L. (2005). Homing endonuclease structure and function. Quarterly Reviews of Biophysics, 1–47.
- Tock, M. R., & Dryden, D. T. F. (2005). The biology of restriction and anti-restriction. Current Opinion in Microbiology, 8, 466–472. https://doi.org/10.1016/j.mib.2005.06.003
- Wilson, G. G., & Murray, N. E. (1991). Restriction and Modification Systems. Annu. Rev. Genet., 25, 585–627.
- Wu, Z., & Mou, K. (2016). Genomic insights into Campylobacter jejuni virulence and population genetics. Infec. Dis. Transl. Med., 2(3), 109–119.
- Yuan, R. (1981). Structure and Mechanism of Multifunctional Restriction Endonucleases. Annu. Rev. Biochem., 50, 285–315.
