ADN polimerasa: tipos, función y estructura
La ADN polimerasa es una enzima que se encarga de catalizar la polimerización de la nueva hebra de ADN durante la replicación de esta molécula. Su función principal es emparejar los desoxirribonucleótidos trifosfato con los de la cadena molde. También participa en la reparación del ADN.
Esta enzima permite el emparejamiento correcto entre las bases de ADN de la cadena molde y la nueva, siguiendo el esquema de A se aparea con T, y G con C.
El proceso de replicación de ADN debe ser efectivo y debe llevarse a cabo con rapidez, por lo que la ADN polimerasa actúa añadiendo unos 700 nucleótidos por segundo y solo comete un error cada 109 o 1010 nucleótidos incorporados.
Existen diferentes tipos de ADN polimerasa. Estas varían tanto en eucariotas como procariotas, y cada una tiene un papel específico en la replicación y reparación del ADN.
Es posible que una de las primeras enzimas en aparecer en la evolución hayan sido las polimerasas, ya que la habilidad para replicar el genoma de forma precisa es un requisito intrínseco para el desarrollo de los organismos.
El descubrimiento de esta enzima se les atribuye a Arthur Kornberg y sus colegas. Este investigador identificó a la ADN polimerasa I (Pol I) en el año 1956, mientras trabaja con Escherichia coli. Del mismo modo, fueron Watson y Crick los que propusieron que esta enzima podía producir copias fieles de la molécula de ADN.
Índice del artículo
Los organismos procariotas (organismos sin un núcleo verdadero, delimitado por una membrana) poseen tres ADN polimerasas principales, abreviadas comúnmente como pol I, II y III.
La ADN polimerasa I participa en la replicación y en la reparación del ADN y posee actividad exonucleasa en ambos sentidos. Se considera que el papel de esta enzima en la replicación es secundario.
La II participa en la reparación del ADN y su actividad exonucleasa es en el sentido 3´-5´. La III participa en la replicación y revisión del ADN, y al igual que la enzima anterior, presenta actividad exonucleasa es en el sentido 3´-5´.
Los eucariotas (organismos con un núcleo verdadero, delimitado por una membrana) poseen cinco ADN polimerasas, denominadas con letras del alfabeto griego: α, β, γ, δ y ε.
La polimerasa γ se localiza en la mitocondria y es la responsable de la replicación del material genético en esta organela celular. En contraste, las otras cuatro se encuentran en el núcleo de las células y están involucradas en la replicación del ADN nuclear.
Las variantes α, δ y ε son las más activas en el proceso de división celular, lo que sugiere que su función principal está asociada con la producción de copias de ADN.
La ADN polimerasa β, por su parte, presenta picos de actividad en las células que no se están dividiendo, por lo que se asume que su función principal se asocia con la reparación del ADN.
Distintos experimentos han logrado comprobar la hipótesis que asocian mayormente a las polimerasas α, δ y ε con la replicación del ADN. Los tipos γ, δ y ε presentan actividad exonucleasa 3´-5´.
Los nuevos métodos de secuenciación han logrado identificar una enorme variedad de familias de ADN polimerasas. En las arqueas, específicamente, se ha identificado una familia de enzimas, denominadas familia D, que son únicas de este grupo de organismos.
El ADN es la molécula que porta toda la información genética de un organismo. Está formada de una azúcar, una base nitrogenada (adenina, guanina, citosina y timina) y un grupo fosfato.
Durante los procesos de división celular, que están ocurriendo constantemente, el ADN debe ser copiado con rapidez y precisión – específicamente en la fase S del ciclo celular. A este proceso donde la célula copia el ADN se le conoce como replicación.
Estructuralmente, la molécula de ADN está formada por dos hebras, formando una hélice. Durante el proceso de replicación, estas se separan y cada una actúa como un templado para la formación de una molécula nueva. Así, las nuevas hebras pasan a las células hijas en el proceso de división celular.
Como cada hebra sirve de templado, se dice que la replicación del ADN es semiconservativa – al final del proceso, la molécula nueva consta de una hebra nueva y una vieja. Este proceso fue descrito en el año 1958 por los investigadores Meselson y Stahl, usando isópotos.
La replicación del ADN requiere de una serie de enzimas que catalizan el proceso. Entre estas moléculas proteicas, resalta la ADN polimerasa.
Para que ocurra la síntesis del ADN, se requieren de los sustratos necesarios para el proceso: los desoxirribonucleótidos trifosfato (dNTP)
El mecanismo de la reacción implica un ataque nucleofílico del grupo hidroxilo en el extremo 3´ de la hebra en crecimiento en el fosfato alfa de los dNTP complementario, eliminando un pirofosfato. Este paso es muy importante, ya que la energía para la polimerización viene de la hidrólisis de los dNTP y del pirofosfato resultante.
La pol III o la alfa se une al primer (ver propiedades de la polimerasas) y empieza a añadir los nucleótidos. La épsilon elonga la cadena líder, y la delta elonga la hebra retardada.
Todas las ADN polimerasas conocidas comparten dos propiedades esenciales asociadas al proceso de replicación.
En primer lugar, todas las polimerasas sintetizan la hebra de ADN en dirección 5´-3´, añadiendo los dNTP al grupo hidroxilo de la cadena en crecimiento.
Segundo, las ADN polimerasas no pueden empezar a sintetizar una cadena nueva de la nada. Necesitan un elemento adicional conocido como primer o cebador, el cual es una molécula formada por unos pocos nucleótidos que otorga un grupo hidroxilo libre, donde la polimerasa puede anclarse y empezar su actividad.
Esta es una de las diferencias fundamentales entre las ADN y ARN polimerasas, ya que esta última si es capaz de iniciar la síntesis de una cadena de novo.
La primera propiedad de las ADN polimerasas mencionada en el apartado anterior, supone una complicación para la replicación semiconservativa. Como las dos hebras de ADN corren de manera antiparalela, una de ellas se sintetiza de manera discontinua (la que necesitaría ser sintetizada en sentido 3´-5´).
En la hebra retardada, la síntesis discontinua ocurre por medio de la actividad normal de la polimerasa, 5´-3´, y los fragmentos resultantes – conocidos en la literatura como fragmentos de Okazaki – son unidos por otra enzima, la ligasa.
El ADN está constantemente expuesto a factores, tanto endógenos como exógenos, que pueden dañarlo. Estos daños pueden bloquear la replicación y acumularse, de manera que afectan la expresión de los genes, generando problemas en los diversos procesos celulares.
Además de su función en el proceso de replicación del ADN, la polimerasa también es un componente clave de los mecanismos de reparación del ADN. También pueden actuar como sensores en el ciclo celular que previenen la entrada a la fase de división si el ADN está dañado.
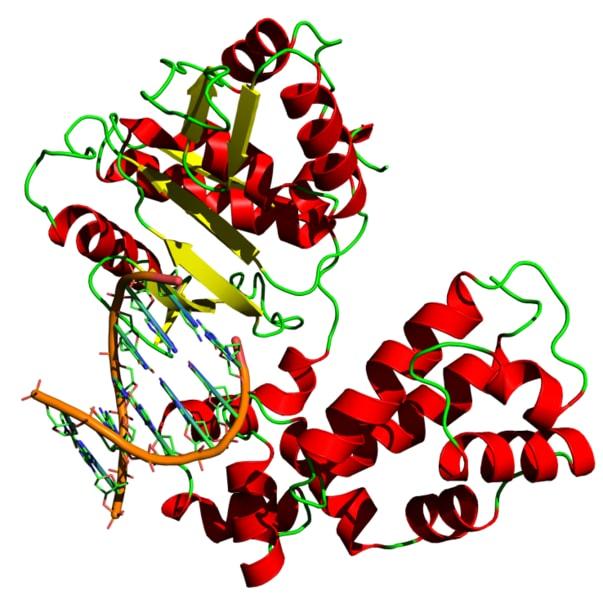
Actualmente, gracias a los estudios de cristalografía, se ha logrado dilucidar las estructuras de diversas polimerasas. Basados en su secuencia primaria, las polimerasas se agrupan en familias: A, B, C, X y Y.
Algunos aspectos son comunes a todas las polimerasas, particularmente los relacionados con los centros catalíticos de la enzima.
Estos incluyen dos sitios activos claves que poseen iones metálicos, con dos residuos de aspartato y un residuo variable – ya sea aspartato o glutamato, que coordina los metales. Existen otra serie de residuos cargados que rodean al centro catalítico y están conservados en las distintas polimerasas.
En procariotas, la ADN polimerasa I es un polipéptido de 103 kd, la II es un polipéptido de 88 kd y la III consta de diez subunidades.
En los eucariotas, las enzimas son más grandes y complejas: la α está formada por cinco unidades, la β y γ por una subunidad, la δ por dos subunidades y.la ε por 5.
La reacción en cadena de la polimerasa (PRC) es un método usado en todos los laboratorios de biología molecular, gracias a su utilidad y simplicidad. El objetivo de este método es amplificar de manera masiva una molécula de ADN de interés.
Para lograrlo, los biólogos usan una ADN polimerasa que no es dañada por el calor (las altas temperatura son indispensables para este proceso) para amplificar la molécula. El resultado de este proceso es un número elevado de moléculas de ADN que pueden ser usadas con distintos propósitos.
Una de las utilidades clínicas más resaltantes de la técnica, es su uso en el diagnóstico médico. La PRC puede ser usada para comprobar la presencia de bacterias y virus patógenos en los pacientes.
Un número importante de medicamentos tienen como objetivo truncar los mecanismos de replicación del ADN en el organismo patógeno, ya sea un virus o una bacteria.
En algunos de esto, el blanco es la inhibición de la actividad de la ADN polimerasa. Por ejemplo, el medicamento quimioterapéutico citarabina, también denominado arabinósido de citosina, inhabilita a la ADN polimerasa.
- Alberts, B., Bray, D., Hopkin, K., Johnson, A. D., Lewis, J., Raff, M., … & Walter, P. (2015). Essential cell biology. Garland Science.
- Cann, I. K., & Ishino, Y. (1999). Archaeal DNA replication: identifying the pieces to solve a puzzle. Genetics, 152(4), 1249-67.
- Cooper, G. M., & Hausman, R. E. (2004). The cell: Molecular approach. Medicinska naklada.
- Garcia-Diaz, M., & Bebenek, K. (2007). Multiple functions of DNA polymerases. Critical reviews in plant sciences, 26(2), 105-122.
- Shcherbakova, P. V., Bebenek, K., & Kunkel, T. A. (2003). Functions of eukaryotic DNA polymerases. Science’s SAGE KE, 2003(8), 3.
- Steitz, T. A. (1999). DNA polymerases: structural diversity and common mechanisms. Journal of Biological Chemistry, 274(25), 17395-17398.
- Wu, S., Beard, W. A., Pedersen, L. G., & Wilson, S. H. (2013). Structural comparison of DNA polymerase architecture suggests a nucleotide gateway to the polymerase active site. Chemical reviews, 114(5), 2759-74.
