Traducción del ADN: proceso en eucariotas y procariotas
La traducción del ADN es el proceso por el cual la información contenida en los ARN mensajeros producidos durante la transcripción (la copia de la información en una secuencia de ADN en forma de ARN) es “traducida” en una secuencia aminoacídica mediante la síntesis proteica.
Desde la perspectiva celular, la expresión de un gen es un asunto relativamente complejo que ocurre en dos pasos: la transcripción y la traducción.
Todos los genes que se expresan (sean o no codificantes para secuencias peptídicas, es decir, proteínas) inicialmente lo hacen mediante la transferencia de la información contenida en su secuencia de ADN a una molécula de ARN mensajero (ARNm) a través de un proceso denominado transcripción.
La transcripción es conseguida por unas enzimas especiales conocidas como ARN polimerasas, que utilizan una de las hebras complementarias del ADN del gen como molde para la síntesis de una molécula de “pre-ARNm”, el cual es procesado posteriormente para formar un ARNm maduro.
Para los genes que codifican proteínas, la información contenida en los ARNm maduros es “leída” y traducida en forma de aminoácidos de acuerdo con el código genético, que especifica qué codón o triplete de nucleótidos corresponde a cada aminoácido en particular.
La especificación de la secuencia aminoacídica de una proteína, por tanto, depende de la secuencia inicial de bases nitrogenadas en el ADN que corresponde al gen y después en el ARNm que transporta dicha información del núcleo al citosol (en las células eucariotas); proceso que también es definido como la síntesis de proteínas guiada por ARNm.
En vista de que existen 64 posibles combinaciones de las 4 bases nitrogenadas que forman el ADN y el ARN y solo 20 aminoácidos, un aminoácido puede ser codificado por distintos tripletes (codones), por lo que se dice que el código genético es “degenerado” (excepto para el aminoácido metionina, que es codificado por un codón único AUG).
Índice del artículo
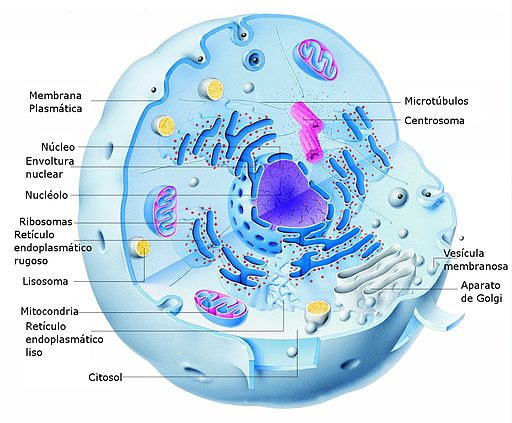
En las células eucariotas la transcripción tiene lugar en núcleo y la traducción en el citosol, por lo que los ARNm que se forman durante el primer proceso cumplen una función también en el transporte de la información desde el núcleo hacia el citosol, donde se encuentran las maquinarias biosintéticas (los ribosomas).
Es importante mencionar que la compartimentación de la transcripción y la traducción en los eucariotas es cierta para el núcleo, pero no es igual para orgánulos con genoma propio como los cloroplastos y las mitocondrias, que tienen sistemas más similares a los de los organismos procariotas.
Las células eucariotas tienen también ribosomas citosólicos unidos a las membranas del retículo endoplásmico (retículo endoplásmico rugoso), en los cuales ocurre la traducción de las proteínas que están destinadas a insertarse en las membranas celulares o que requieren de los procesamientos postraduccionales que ocurren en dicho compartimento.
Los ARNm son modificados en sus extremos a medida que son transcritos:
– Cuando el extremo 5’ del ARNm emerge de la superficie de la ARN polimerasa II durante la transcripción, este es “atacado” inmediatamente por un grupo de enzimas que sintetizan una “capucha” compuesta por 7-metil guanilato y que está conectada al nucleótido terminal del ARNm a través de un enlace trifosfato 5’,5’.
– El extremo 3’ del ARNm sufre un “clivaje” por una endonucleasa, lo que genera un grupo hidroxilo libre 3’ al cual se une una “ristra” o “cola” de residuos de adenina (de 100 a 250) que son añadidos uno a la vez por una enzima poly (A) polimerasa.
La “capucha 5’” y la “cola poly A” cumplen funciones en la protección de las moléculas de ARNm frente a la degradación y, además, funcionan en el transporte de los transcritos maduros hacia el citosol y en la iniciación y terminación de la traducción, respectivamente.
Corte y empalme
Tras la transcripción, los ARNm “primarios” con sus dos extremos modificados, aún presentes en el núcleo, pasan por un proceso de “corte y empalme” mediante el cual generalmente se eliminan las secuencias intrónicas y se unen los exones resultantes (procesamiento postranscripcional), con lo que se obtienen los transcritos maduros que abandonan el núcleo y llegan al citosol.
El corte y empalme es llevado a cabo por un complejo riboproteico denominado el espliceosoma (anglicismo de spliceosoma), formado por cinco ribonucleoproteínas pequeñas y moléculas de ARN, que son capaces de “reconocer” las regiones que deben eliminarse del transcrito primario.
En muchos eucariotas se da un fenómeno conocido como “corte y empalme alternativo”, lo que significa que distintos tipos de modificaciones postranscripcionales pueden producir proteínas diferentes o isoenzimas que difieren entre sí en algunos aspectos de sus secuencias.
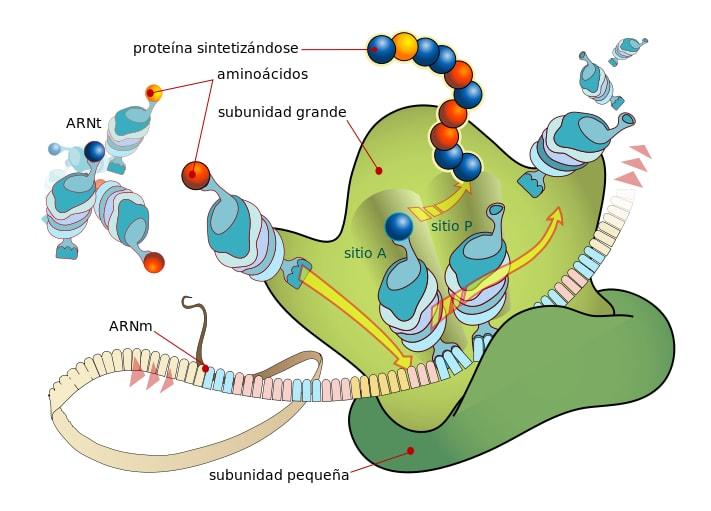
Cuando los transcritos maduros abandonan el núcleo y son transportados para su traducción en el citosol, estos son procesados por el complejo traduccional conocido como ribosoma, que consiste en un complejo de proteínas asociadas con moléculas de ARN.
Los ribosomas están compuestos por dos subunidades, una “grande” y otra “pequeña”, las cuales están disociadas libremente en el citosol y se unen o asocian sobre la molécula de ARNm que se traduce.
La unión entre los ribosomas y el ARNm depende de unas moléculas de ARN especializadas que se asocian con las proteínas ribosomales (ARN ribosomal o ARNr y ARN de transferencia o ARNt), cada una de las cuales ejerce funciones específicas.
Los ARNt son “adaptadores” moleculares, pues a través de uno de sus extremos pueden “leer” cada codón o triplete en el ARNm maduro (por complementariedad de bases) y a través del otro pueden unirse al aminoácido codificado por el codón “leído”.
Las moléculas de ARNr, por otro lado, se encargan de acelerar (catalizar) el proceso de unión de cada aminoácido en la cadena peptídica naciente.
Un ARNm maduro eucariota puede ser “leído” por muchos ribosomas, tantas veces como la célula lo indique. En otras palabras, el mismo ARNm puede dar lugar a muchas copias de la misma proteína.
Codón de inicio y marco de lectura
Cuando un ARNm maduro es abordado por las subunidades ribosomales, el complejo riboproteico “escanea” la secuencia de dicha molécula hasta que encuentra un codón de inicio, que siempre es AUG e implica la introducción de un residuo de metionina.
El codón AUG define el marco de lectura para cada gen y, además, define el primer aminoácido de todas las proteínas traducidas en la naturaleza (este aminoácido muchas veces es eliminado postraduccionalmente).
Codones de terminación
Otros tres codones han sido identificados como aquellos que inducen la terminación de la traducción: UAA, UAG y UGA.
Aquellas mutaciones que implican un cambio de bases nitrogenadas en el triplete que codifica para un aminoácido y que resultan en codones de terminación se conocen como mutaciones sin sentido, pues provocan una detención prematura del proceso de síntesis, lo que forma proteínas más cortas.
Regiones no traducidas
Cerca del extremo 5’ de las moléculas de ARNm maduras existen regiones que no son traducidas (UTR, del inglés UnTranslated Region), también denominadas secuencias “líder”, las cuales están ubicadas entre el primer nucleótido y el codón de inicio de la traducción (AUG).
Estas regiones UTR que no se traducen tienen sitios específicos para la unión con ribosomas y en los humanos, por ejemplo, tienen una longitud aproximada de 170 nucleótidos, entre los cuales existen regiones reguladoras, sitios de unión de proteínas que funcionan en la regulación de la traducción, etc.
La traducción, así como la transcripción consiste en 3 fases: una de iniciación, otra de elongación y finalmente una de terminación.
Iniciación
Consiste en el ensamblaje del complejo traduccional sobre el ARNm, lo que amerita la unión de tres proteínas conocidas como factores de iniciación (IF, del inglés Initiation Factor) IF1, IF2 e IF3 a la subunidad pequeña del ribosoma.
El complejo de “preiniciación” formado por los factores de iniciación y la subunidad ribosomal pequeña se unen, a su vez, con un ARNt que “carga” un residuo de metionina y este conjunto de moléculas se une al ARNm, cerca del codón de inicio AUG.
Estos eventos conducen a la unión del ARNm con la subunidad ribosomal grande, lo que conlleva a la liberación de los factores de iniciación. La subunidad grande del ribosoma tiene 3 sitios de unión para moléculas de ARNt: el sitio A (aminoácido), el sitio P (polipéptido) y el sitio E (de salida).
El sitio A se une al anticodón del aminoacil-ARNt que es complementario con el del ARNm que se traduce; el sitio P es donde el aminoácido es transferido desde el ARNt al péptido naciente y el sitio E es donde se encuentra en ARNt “vacío” antes de ser liberado al citosol después de entregar el aminoácido.
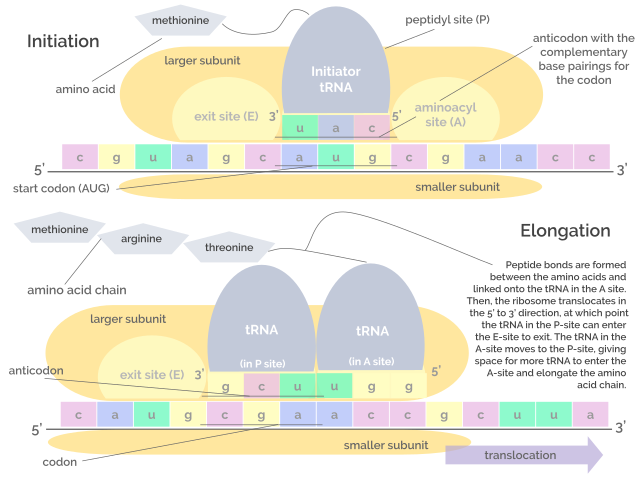
Elongación
Esta fase consiste en el “movimiento” del ribosoma a lo largo de la molécula de ARNm y de la traducción de cada codón que va “leyendo” lo que implica el crecimiento o la elongación de la cadena polipeptídica en nacimiento.
Este proceso requiere de un factor conocido como factor de elongación G y de energía en forma de GTP, que es la que impulsa la translocación de los factores de elongación a lo largo de la molécula de ARNm mientras esta se va traduciendo.
La actividad peptidil transferasa de los ARN ribosomales permite la formación de los enlaces peptídicos entre los aminoácidos sucesivos que se adicionan a la cadena.
Terminación
La traducción finaliza cuando el ribosoma se encuentra con alguno de los codones de terminación, pues los ARNt no reconocen a estos codones (no codifican aminoácidos). También se unen proteínas conocidas como factores de liberación, los cuales facilitan el desprendimiento del ARNm del ribosoma y la disociación de las subunidades de este.
En los procariotas, al igual que en las células eucariotas, los ribosomas encargados de la síntesis de proteínas se encuentran en el citosol (lo que también es cierto para la maquinaria transcripcional), hecho que permite el rápido incremento de la concentración citosólica de una proteína cuando aumenta la expresión de los genes que la codifican.
Aunque no es un proceso sumamente común en estos organismos, los ARNm primarios producidos durante la transcripción pueden sufrir maduración postranscripcional a través de “corte y empalme”. No obstante, lo más común es observar ribosomas unidos al transcrito primario que lo van traduciendo al mismo tiempo que este va siendo transcrito desde la secuencia de ADN correspondiente.
En vista de lo anterior, la traducción en muchos procariotas comienza por el extremo 5’, puesto que el extremo 3’ del ARNm permanece unido al ADN molde (y ocurre concomitantemente con la transcripción).
Regiones no traducidas
Las células procariotas también producen ARNm con regiones no traducidas que se conocen como la “caja Shine-Dalgarno” y cuya secuencia consenso es AGGAGG. Como es evidente, las regiones UTR de las bacterias son considerablemente más cortas que las de las células eucariotas, aunque ejercen funciones similares durante la traducción.
En las bacterias y los demás organismos procariotas el proceso de traducción es bastante parecido al de las células eucariotas. Consiste también en tres fases: iniciación, elongación y terminación, las cuales dependen de factores procariotas específicos, distintos a los que emplean los eucariotas.
La elongación, por ejemplo, depende de factores de elongación conocidos como EF-Tu y EF-Ts, en vez del factor G de los eucariotas.
- Alberts, B., Johnson, A., Lewis, J., Raff, M., Roberts, K., & Walter, P. (2007). Molecular biology of the cell. Garland Science. New York, 1392.
- Clancy, S. & Brown, W. (2008) Translation: DNA to mRNA to Protein. Nature Education 1(1):101.
- Griffiths, A. J., Wessler, S. R., Lewontin, R. C., Gelbart, W. M., Suzuki, D. T., & Miller, J. H. (2005). An introduction to genetic analysis. Macmillan.
- Lodish, H., Berk, A., Kaiser, C. A., Krieger, M., Scott, M. P., Bretscher, A., … & Matsudaira, P. (2008). Molecular cell biology. Macmillan.
- Nelson, D. L., Lehninger, A. L., & Cox, M. M. (2008). Lehninger principles of biochemistry. Macmillan.
- Rosenberg, L. E., & Rosenberg, D. D. (2012). Human Genes and Genomes: Science. Health, Society, 317-338.
