Enolasa: estructura, mecanismo de acción, funciones
La enolasa es la enzima encargada de llevar a cabo la conversión de D-2-fosfoglicerato (2PGA) en fosfoenolpiruvato (PEP) en la glicólisis y la reacción reversa en la gluconeogénesis, dos rutas metabólicas que forman parte del metabolismo energético celular.
La decisión de catalizar esta reacción en una u otra dirección depende del acceso que tenga la célula a la glucosa. Es decir, de las necesidades que tenga de adaptar su metabolismo a la degradación o a la síntesis a fin de obtener energía. Indispensable para la realización de sus procesos vitales.
Dado que ambas rutas metabólicas pertenecen al centro del árbol metabólico central de los seres vivos, no resulta extraño que la secuencia de aminoácidos de esta proteína se halle conservada en archaeas, bacterias y eucariotas. Y por ende de que presente propiedades catalíticas similares.
La localización de la enolasa en la célula se limita al citosol, compartimiento en el que tienen lugar tanto la glicólisis (denominada también glucólisis) como la gluconeogénesis en la mayoría de los organismos.
No obstante, ha sido también detectada en otros compartimientos celulares tales como membrana plasmática de muchos patógenos y células cancerosas. Allí, parece estar involucrada con la facilitación de procesos de diseminación celular, una función totalmente distinta a su función clásica.
Las enzimas capaces de realizar más de una función, tal y como lo hace la enolasa, son conocidas como enzimas moonlighting.
Índice del artículo
- 1 Estructura
- 2 Mecanismo de acción
- 3 Funciones
- 4 La enolasa y su relación con los mecanismos de diseminación celular
- 5 Referencias
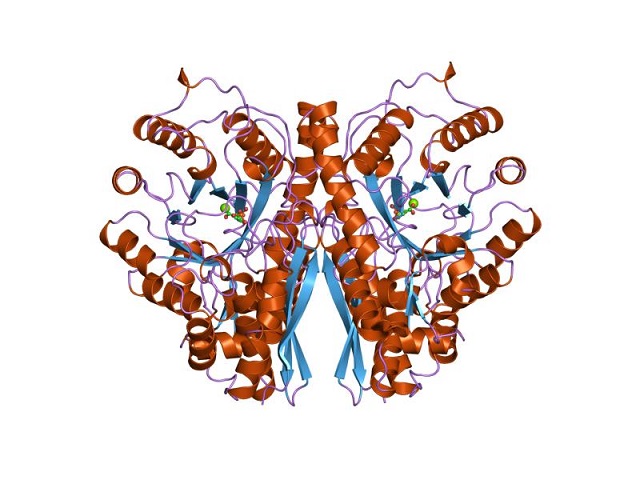
La estructura cuaternaria de la enolasa unida o no a sus ligandos ha sido determinada en un gran número de individuos procariotas y eucariotas.
Cada monómero presenta dos dominios: un pequeño dominio amino-terminal y un dominio carboxilo-terminal de mayor tamaño. El dominio N-terminal se encuentra integrado por tres hélices α y cuatro láminas β. Mientras que, el C-terminal se compone de ocho laminas β que se alternan entre ellas formando un β-barril que se encuentra rodeado por ocho hélices α.
Además, dos sitios de unión para cationes divalentes se encuentran en cada monómero los cuales se han denominados “sitio conformacional” y “sitio catalítico”. El primero, es poco selectivo y puede unir una gran variedad de cationes divalentes en ausencia de sustrato.
Mientras que, el segundo se une a los iones luego de que el sustrato se ha unido a la enzima. La unión de iones a ambos sitios es vital para que la reacción pueda proceder.
Finalmente, resulta importante mencionar que en los homodímeros, los monómeros se unen manteniendo una orientación paralela. Por tanto, el sitio activo queda limitado a la región central formada por dicha unión.
No obstante, en la catálisis solo participan residuos de uno solo de los dos monómeros. Esto explica que la capacidad de los monómeros de llevar a cabo la reacción bajo condiciones experimentales.
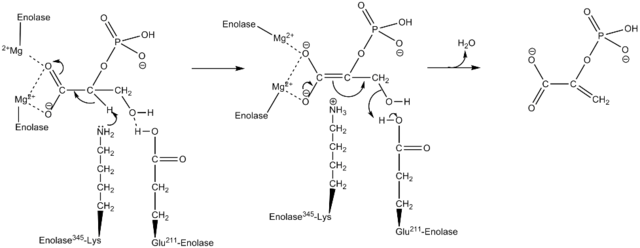
Los estudios estructurales, así como aquellos que han permitido determinar las características cinéticas y fisicoquímicas de la enolasa, han permitido entender su mecanismo de acción.
La manera en la que la enzima lleva a cabo la catálisis de la reacción resulta bastante interesante. A pesar de que, un solo sustrato se encuentra involucrado, un mecanismo secuencial ordenado es el que se ha propuesto.
Este comienza con la unión de un ión de Mg2+ al sitio conformacional de uno de los monómeros. Continua con la unión del sustrato al sitio activo seguida de la unión de un segundo ión al sitio catalítico y concluye con la pronta liberación del producto una vez que ha sido llevada a cabo la reacción. En este punto, el Mg2+ permanece unido al sitio conformacional.
En esta misma línea, para favorecer la realización de la reacción la enzima media en primera instancia la generación de un intermediario carbanión, eliminando un protón del carbono 2 del 2PGA. Esto lo hace gracias a la acción de un residuo aminoacídico básico.
Secuencialmente, tiene lugar la eliminación del hidroxilo del carbono 3 por la acción de un residuo ácido de la enzima. En este punto, se lleva a cabo la unión de ambos carbonos mediante un doble enlace formando PEP. De este modo se da por culminada la reacción.
Muchas de las enzimas hasta ahora estudiadas son capaces de realizar una gran variedad de funciones no relacionadas a su “función clásica” en compartimientos celulares diferentes. Estas enzimas han sido denominadas como enzimas “moonlighting”.
En tal sentido, la enolasa puede ser considerada como una enzima moonlighting, ya que numerosas funciones opuestas a su función clásica le han sido hasta la fecha atribuidas tanto en bacterias como en eucariotas.
Algunas de estas funciones son las siguientes:
– Participa en el mantenimiento de la forma celular así como en el tráfico vesicular al interactuar con proteínas del citoesqueleto.
– En el núcleo de las células de mamífero actúa como factor de transcripción que regula la expresión de genes asociados con la proliferación celular. Coopera en el mantenimiento de la estabilidad de los ARNm en el degradosoma en bacterias.
– En los patógenos, tales como Streptococcus pneumoniae y Trypanosoma cruzi, parece que actúa como un importante factor de virulencia.
– También se ha encontrado que en Streptococcus pyogenes, la enolasa se excreta al medio extracelular facilitando la degradación de tejidos y la evasión del sistema inmune en el hospedador.
– En la superficie de células tumorales es expresada potenciando la metástasis.
Numerosos patógenos, así como células tumorales, expresan en su membrana o excretan al medio extracelular proteasas capaces de degradar proteínas de la matriz extracelular.
Esta capacidad le permite a dichas células abrirse paso entre los tejidos y diseminarse rápidamente a lo largo de todo el organismo hospedador. Favoreciendo de esta manera la evasión del sistema inmune y por tanto, el establecimiento de la infección.
Aun cuando la enolasa carece de actividad proteasa, participa en el proceso de diseminación de muchos patogenos en su hospedador así como de células tumorales durante la metastasis.
Esto lo logra gracias a que se expresa en la superficie de estas células funcionando como receptor de plasminógeno. Este último, es el zimógeno de una serin-proteasa conocida como plasmina que forma parte del sistema fibrinolítico y actúa degradando proteínas de matriz extracelular.
Por tanto, la enolasa expresada en la superficie es una estrategia que han adquirido estas células para establecer la infección y diseminarse de manera exitosa.
Dicha estrategia consiste de dos procesos:
– La evasión del sistema inmune del hospedador. Ya que, al recubrirse estas células de una proteína propia del hospedador son ignoradas por las células del sistema inmune que reconocen proteínas no propias asociadas a patógenos.
– La diseminación post-activación del plasminógeno en plasmina. Cuya participación en la degradación de proteínas de matriz extracelular, facilita entonces la rápida y efectiva diseminación.
- Avilan L, Gualdron-Lopez M, Quiñones W, González-González L, Hannaert V, Michels PAA, Concepción JL. Enolase: a key player in the metabolism and a probable virulence factor of trypanosomatid parasites-perspectives for its use as a therapeutic target. Enzyme Research. 2011 vol. Article ID932549, 14 pages.
- Bhowmick I, Kumar N, Sharma S, Coppens I, Jarori GK, Plasmodium falciparum enolase: stage-specific expression and sub-cellular localization. Malaria Journal. 2009; 8(1). article 179.
- Day I, Peshavaria M, Quinn GB, A differential molecular clock in enolase isoprotein evolution. Journal of Molecular Evolution. 1993; 36(6):599-601.
- de la Torre-Escudero E, Manzano-Román R, Pérez-Sánchez R, Siles-Lucas M, Oleaga A. Cloning and characterization of a plasminogen-binding surface-associated enolase from Schistosoma bovis. Veterinary Parasitology. 2010; 173:73-84.
- Dinovo EC, Boyer PD. Isotopic probes of the enolase reaction mechanism. Initial and equilibrium isotope exchange rates: primary and secondary isotope effects. J Biol Chem. 1971; 246 (14):4586-4593.
- Kaberdin VR, Lin-Chao S, Unraveling new roles for minor components of the E. coli RNA degradosome. RNA Biology. 2009; 6(4):402-405.
- Keller A, Peltzer J, Carpentier G. Interactions of enolase isoforms with tubulin and microtubules during myogenesis. Biochimica et Biophysica Acta. 2007; 1770(6):919-926.
- Lung J, Liu KJ, Chang JY, Leu SJ, Shih NY. MBP-1 is efficiently encoded by an alternative transcript of the ENO1 gene but post-translationally regulated by proteasome-dependent protein turnover. FEBS Journal. 2010; 277(20):4308-4321.
- Pancholi V. Multifunctional α-enolase: its role in diseases. Cellular and Molecular Life Sciences. 2001; 58(7):902-920.
- Poyner RR, Cleland WW, Reed GH. Role of metal ions in catalysis by enolase. An ordered kinetic mechanism for a single substrate enzyme. Biochemistry. 2001; 40:9008-8017.
- Segovia-Gamboa NC, Chávez-Munguía B, Medina-Flores A, Entamoeba invadens, encystation process and enolase. Experimental Parasitology. 2010; 125(2):63-69.
- Tanaka M, Sugisaki K, Nakashima K, Switching in levels of translatable mRNAs for enolase isozymes during development of chicken skeletal muscle. Biochemical and Biophysical Research Communications. 1985; 133(3):868-872.
