Replicación del ADN: mecanismos, en procariotas y eucariotas
La replicación del ADN (ácido desoxirribonucleico) consiste en copiar el genoma, es decir, toda la información genética contenida en el ADN de un organismo, para producir dos copias idénticas. El genoma posee la información necesaria para construir un organismo completo.
Antes de la división celular, ocurre la replicación del ADN. Mediante la meiosis, se producen gametos para la reproducción sexual. Mediante la mitosis, se produce el reemplazo de células (por ejemplo, de la piel y la sangre) y el desarrollo (por ejemplo, de tejidos y órganos).
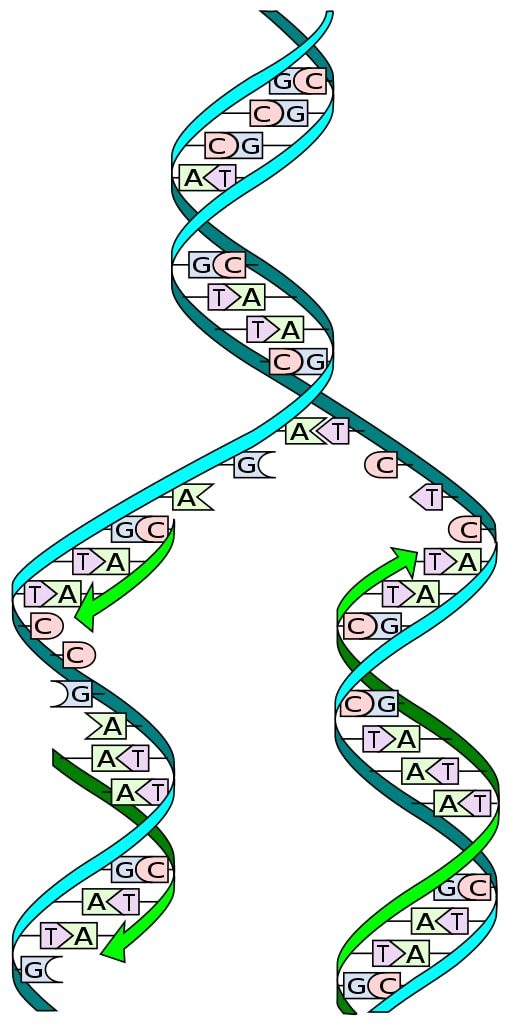
El conocimiento de la estructura del ADN permite entender la manera en que ocurre su replicación. La estructura del ADN consiste en una doble hélice, compuesta por dos cadenas antiparalelas de nucleótidos sucesivos, cuyas bases nitrogenadas se complementan de forma específica.
Durante la replicación, cada una de las cadenas de la doble cadena de ADN actúa como molde para la biosíntesis de una nueva cadena. Las dos cadenas recién sintetizadas tienen bases que son complementarias de las bases de la cadena molde: adenina (A) con timina (T), y citosina (C) con guanina (G).
Varias enzimas y proteínas participan en la replicación del ADN. Por ejemplo, abriendo la doble hélice de ADN, manteniendo el ADN abierto, y adicionando desoxiribonucleosidos-5′-trifosfato (dNTP) para formar la cadena nueva.
Índice del artículo
- 1 La replicación del ADN es semiconservativa
- 2 Replicación en baterias
- 3 Mecanismos que aseguran la fidelidad de replicación del ADN
- 4 Replicación del ADN en eucariotas
- 5 La replicación del ADN en eucariotas y el ciclo celular
- 6 Replicación de los extremos de los cromosomas en los eucariotas
- 7 Las funciones de otras ADN polimerasas en los eucariotas
- 8 La replicación del ADN en arqueobacterias
- 9 Referencias
Basándose en la estructura del ADN, Watson y Crick propusieron que la replicación del ADN ocurre de forma semiconservativa. Esto fue demostrado por Meselson y Stahl mediante el marcaje del ADN de Escherichia coli con el isotopo pesado del nitrógeno, 15N, siguiendo por varias generaciones el patrón de distribución en un medio de cultivo con nitrógeno liviano, 14N.
Meselson y Stahl encontraron que, en la primera generación, las dos moléculas de ADN hijas tenían, cada molécula marcada con una cadena con el isótopo pesado de nitrógeno y otra con el isotopo liviano. A diferencia de la molécula de ADN parental, que tenía las dos cadenas marcadas con el isotopo pesado, 15N.
En la segunda generación, el 50% de las moléculas de ADN eran como las de la primera generación, y el otro 50% tenían únicamente nitrógeno liviano. La interpretación de este resultado es que la doble hélice hija tienen una cadena parental (que funciona como molde) y una cadena nueva.
El mecanismo de replicación semiconservativa implica la separación de las cadenas del ADN y apareamiento de bases complementaria mediante el emparejamiento de nucleótidos sucesivos, produciendo dos dobles hélices hijas.
El ADN de las bacterias consiste de un cromosoma circular y tiene solo un sitio de origen de la replicación. A partir de este sitio, la biosíntesis de las dos cadenas hijas sucede bidireccionalmente, formándose dos horquillas de replicación que se mueven en direcciones opuestas al origen. Al final, las horquillas se encuentran, completando la replicación.
La replicación comienza con la unión de las proteínas DnaA al sitio de origen. Estas proteínas a su vez forman un complejo. Luego se unen las proteínas HU e IHF, entre otras, que en conjunto doblan el ADN, ocasionando la separación de las dos cadenas de ADN en una región rica en timina y adenina.
Seguidamente, se unen proteínas DNaC, las cuales hacen que se unan las helicasas del ADN. Estas ayudan a desenrollar el ADN y a romper los enlaces de hidrógeno, formados entre pares de bases. Entonces, las dos cadenas se separan aún más, formando dos cadenas simples.
La topoisomerasa II, o girasa del ADN, se mueve en frente de la helicasa del ADN disminuyendo los superenrollamientos positivos. Las proteínas de unión al ADN de una sola cadena (SSB) mantienen las cadenas del ADN separadas. Así, la biosíntesis de la cadena hija puede comenzar.
La enzima primasa se encarga de sintetizar cadenas cortas de ARN llamadas cebadores, las cuales tienen de 10 a 15 nucleótidos de longitud. La ADN polimerasa comienza a agregar desoxinucleósidos 5′-trifosfato (dNTPs) al extremo 3′-OH del azúcar del cebador, tras lo cual la cadena continúa creciendo por el mismo extremo.
Debido a que las cadenas del ADN son antiparalelas, un cebador es sintetizado en la cadena guía y muchos cebadores en la cadena retardada. Debido a ello, la biosíntesis de la cadena retardada es discontinua. Pese a que las cadenas del ADN son antiparalelas, la horquilla de replicación se mueve en una sola dirección.
La ADN polimerasa se encarga de la formación de enlaces covalentes entre nucleótidos adyacentes de las cadenas recién sintetizadas, en dirección 5’®3′. En E. coli, hay cinco ADN polimerasas: las ADN polimerasas I y III realizan la replicación del ADN; y las ADN polimerasas II, IV y V se encargan de reparar y replicar el ADN dañado.
La mayor parte de la replicación es realizada por la ADN polimerasa III, la cual es una holoenzima que posee 10 subunidades diferentes con varias funciones en la replicación del ADN. Por ejemplo, la subunidad alfa se encarga de realizar enlaces entre nucleótidos.
La helicasa del ADN y la primasa se unen para formar un complejo denominado primosoma. Este se mueve a lo largo del ADN, actuando de forma coordinada para separar las dos cadenas parentales, sintetizando los cebadores cada cierto intervalo sobre la cadena retardada.
El primosoma se une físicamente a la ADN polimerasa III, y forma el replisoma. Dos ADN polimerasas III se encargan de replicar el ADN de las cadenas guía y retardada. Con respecto a la ADN polimerasa III, la cadena retardada forma un lazo hacia afuera, lo cual permite que la adición de nucleótidos a esta cadena suceda en la misma dirección de la cadena guía.
La adición de nucleótidos a la cadena guía es continua. Mientras que en la retardada es discontinua. Se forman fragmentos de 150 nucleótidos de longitud, denominados fragmentos de Okazaki.
La actividad de exonucleasa 5′ -> 3′ de la ADN polimerasa I se encarga de eliminar los cebadores y de rellenar, agregando nucleótidos. Una enzima ligasa sella los huecos entre fragmentos. La replicación termina cuando las dos hoquillas de replicación se encuentran en una secuencia de terminación.
La proteína Tus se une a la secuencia de terminación, deteniendo el movimiento de la horquilla de replicación. La topoisomerasa II permite la separación de los dos cromosomas.
El desoxinucleósido trifosfato (dNTP) contiene tres grupos fosfato unidos al carbono 5′ de la desoxiribosa. Los dNTPs (dATP, dTTP, dGTP y dCTP) se unen a la cadena molde siguiendo la regla AT/GC.
La ADN polimerasa cataliza la reacción siguiente: el grupo hidroxilo (–OH) 3′ del nucleótido de la cadena creciente reacciona con el fosfato alfa del dNTP entrante, liberando pirofosfato inorgánico (PPi). La hidrólisis de PPi produce la energía para la formación del enlace covalente, o enlace fosfodiéster, entre nucleótidos de la cadena creciente.
Durante la replicación del ADN, la ADN polimerasa III comete un error por 100 millones de nucleótidos. Aunque la probabilidad de error es muy baja, hay mecanismos que aseguran la fidelidad en la replicación del ADN. Estos mecanismos son:
1) Estabilidad en el apareamiento de bases. La energía de enlace de hidrógeno entre AT/GC es mayor que en pares de bases erróneos.
2) Estructura del sitio activo de la ADN polimerasa. La ADN polimerasa cataliza preferencialmente uniones de nucleótidos con bases correctas en la cadena opuesta. Un mal apareamiento de bases ocasiona una distorsión de la doble hélice de ADN, que evita que el nucleótido incorrecto ocupe el sitio activo de la enzima.
3) Prueba de lectura. La ADN polimerasa identifica los nucleótidos erróneos incorporados y los elimina de la cadena hija. La actividad exonucleasa de la ADN polimerasa rompe los enlaces fosfodiéster entre nucleótidos en el extremo 3′ de la cadena nueva.
A diferencia de la replicación en procariotas, cuya replicación comienza en un solo sitio, la replicación en eucariotas comienza en múltiples sitios de origen y la horquilla de replicación se mueve bidireccionalmente. Posteriormente, todas las horquillas de replicación se fusionan, formándose dos cromátidas hermanas unidas en el centrómero.
Los eucariotas poseen muchos tipos de ADN polimerasa, cuyos nombres usan letras griegas. La ADN polimerasa α forma un complejo con la primasa. Este complejo sintetiza cebadores cortos que constan de 10 nucleótidos de ARN seguidos de 20 a 30 nucleótidos de ADN.
Luego, la ADN polimerasa ε o δ cataliza la elongación de la cadena hija a partir del cebador. La ADN polimerasa ε está implicada en la síntesis de la cadena líder, mientras que la ADN polimerasa δ sintetiza la cadena retardada.
La ADN polimerasa δ alarga el fragmento de Okazaki de la izquierda hasta alcanzar el cebador de ARN de la derecha, produciendo un levantamiento corto del cebador (“short flap”). A diferencia de los procariotas, donde una ADN polimerasa elimina el cebador, en los eucariotas una enzima endonucleasa Flap elimina el cebador de ARN.
A continuación, una ligasa del ADN sella los fragmentos de ADN que se encuentran adyacentes. La terminación de la replicación tiene lugar con la disociación de proteínas de la horquilla de replicación.
La replicación en eucariotas ocurre en la fase S del ciclo celular. Las moléculas de ADN replicadas son segregadas en dos células hijas durante la mitosis. Las fases G1 y G2 separan la fase S y la mitosis. La progresión a través de cada fase del ciclo celular está altamente regulada por quinasas, fosfatasas y proteasas.
En la fase G1 del ciclo celular, el complejo de reconocimiento del origen (OCR) se une al sitio de origen. Esto induce la unión de helicasas MCM y otras proteínas, como Cdc6 y Cdt1, para que formen un complejo de pre-replicación (preRC). Las helicasa MCM se une a la cadena guía.
En la fase S, preRC se convierte en un sitio de replicación activo. Las proteínas OCR, Cdc6 y Cdt1 son liberadas, y la helicasa MCM se mueve en la dirección del 3′ a la 5′. Una vez que termina la replicación, esta será reiniciada en el siguiente ciclo celular.
A los extremos de los cromosomas se les conoce como telómeros, los cuales consisten en secuencias repetidas en tándem, y de una región 3′ que sobresale, de 12 a 16 nucleótidos de longitud.
La ADN polimerasa es incapaz de replicar el extremo 3′ de las cadenas de ADN. Ello se debe a que la DNA polimerasa sólo puede sintetizar ADN en la dirección 5′-3′, y solo puede alargar cadenas preexistentes, sin poder sintetizar un cebador en esta región. En consecuencia, los telómeros se acortan en cada ronda de replicación.
La enzima telomerasa evita el acortamiento de los telómeros. La telomerasa es una enzima que posee subunidades de proteína y ARN (TERC). Este último se une a las secuencias repetidas del ADN, y permite que la telomerasa se una al extremo 3′ del telómero.
Una secuencia de ARN detrás del sito de unión funciona como molde para la síntesis de una secuencia de seis nucleótidos (polimerización) en el extremo de la cadena de ADN. El alargamiento del telómero es catalizado por subunidades de la telomerasa, denominados transcriptasa inversa de la telomerasa (TERT).
Después de la polimerización, tiene lugar la translocalización, consistente en el movimiento de la telomerasa a un nuevo extremo de la cadena de ADN, uniendo otros seis nucleótidos hasta el final.
La ADN polimerasa β tiene un papel importante en la eliminación de bases incorrectas del ADN, pero no está implicada en la replicación del ADN.
Muchas ADN polimerasas descubiertas pertenecen al grupo de las polimerasas “translesion-replicating”. Estas polimerasas se encargan de sintetizar cadenas complementarias en una región de ADN dañado.
Hay varios tipos de polimerasas “translesion-replicating”. Por ejemplo, la ADN polimerasa η puede replicar sobre dímeros de timina, que son producidos por luz UV.
La replicación del ADN de arqueobacterias es similar a la que se da en eucariotas. Ello se debe a lo siguiente: 1) las proteínas que participan en la replicación se parecen más a las de eucariotas que a las de procariotas; y 2) aunque solo hay un sitio de replicación tal como en los procariotas, su secuencia es similar al sitio de origen de eucariotas.
La similitud en la replicación entre los Arquea y los eucariotas apoya la idea de que ambos grupos están filogenéticamente más relacionados entre sí que cualquiera de ambos con los procariotas.
- Brooker, R. J. 2018. Genetics analysis and principles. McGraw-Hill, Nueva York.
- Hartwell, L. H., Goldberg, M. L., Fischer, J. A., Hood, L. 2018. Genetics – from genes to genomes. McGraw-Hill, Nueva York.
- Kušić-Tišma, J. 2011. Fundamental aspects of DNA replication. InTech Open access, Croacia.
- Lewis, R., 2015. Human genetics concepts and applications. McGraw-Hill, Nueva York.
- Pierce, B. A. 2005. Genetics – a conceptual approach. W. H. Freeman, Nueva York.
