¿Qué es el empaquetamiento del ADN?
El empaquetamiento del ADN es un término que define la compactación controlada del ADN en el interior celular. En ninguna célula (y de hecho, ni siquiera en los virus) el ADN está libre, laxo y en solución verdadera.
El ADN es una molécula extremadamente larga que, además, está siempre interactuando con una enorme variedad de distintas proteínas. Para el procesamiento, herencia y control de la expresión de los genes que porta, el ADN adopta una organización espacial particular. Esto lo consigue la célula controlando estrictamente cada paso del empaquetamiento del ADN a distintos niveles de compactación.
Los virus poseen distintas estrategias de empaquetamiento de sus ácidos nucleicos. Una de las favoritas es la de la formación de espirales compactas. Podría decirse que los virus son ácidos nucleicos empaquetados en las propias proteínas que los cubren, protegen y movilizan.
En procariotas, el ADN está asociado a proteínas que determinan la formación de lazos complejos en una estructura que se denomina nucleoide. El nivel máximo de compactación del ADN en una célula eucariota, por otro lado, es el cromosoma mitótico o meiótico.
La única instancia en la cual un ADN-B no está empaquetado es un laboratorio de investigación que persigue tal fin.
Índice del artículo
- 1 Estructura del ADN
- 2 El nucleoide bacteriano
- 3 Los niveles de compactación del cromosoma eucariota
- 4 La compactación meiótica del ADN
- 5 Referencias
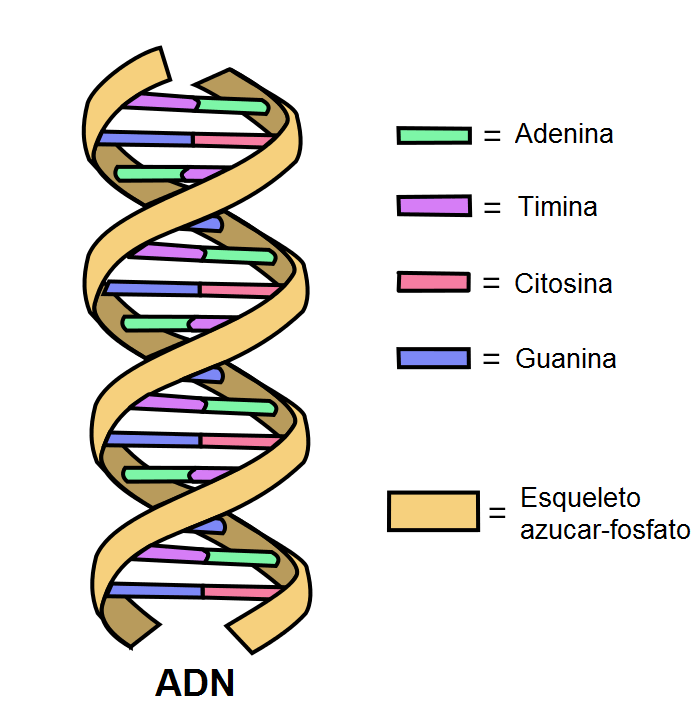
El ADN está formado por dos bandas antiparalelas que forman una hélice doble. Cada una de ellas presenta un esqueleto de enlaces fosfodiéster sobre los cuales se unen azúcares ligadas a bases nitrogenadas.
En el interior de la molécula, las bases nitrogenadas de una banda forman puentes de hidrógeno (dos o tres) con la banda complementaria.
En una molécula como esta, la mayoría de los ángulos de enlace importantes muestran libre rotación. Los enlaces base nitrogenada-azúcar, azúcar-grupo fosfato y enlace fosfodiéster son flexibles.
Esto permite que el ADN, visto como una varilla flexible, muestre cierta capacidad para doblarse y para enrollarse. Esta flexibilidad permite al ADN adoptar estructuras locales complejas, y formar lazos de interacción a corta, mediana y larga distancia.
Esta flexibilidad explica también cómo se pueden mantener 2 metros de ADN en cada célula diploide de un ser humano. En un gameto (célula haploide), sería un metro de ADN.
Aunque no sea una regla inquebrantable, el cromosoma bacteriano existe como una única molécula de ADN doble banda superenrollada.
La doble hélice se tuerce más sobre sí misma (más de 10 pb por vuelta) produciendo así cierto compactamiento. Se generan también nudos locales gracias a manipulaciones que están controladas enzimáticamente.
Además, existen secuencias en el ADN que permiten que se formen dominios en grandes lazos. A la estructura resultante del supererollamiento y de lazos ordenados lo llamamos nucleoide.
Estos experimentan cambios dinámicos gracias a algunas proteínas que le brindan cierta estabilidad estructural al cromosoma compactado. El grado de compactación en bacterias y arqueas es tan eficiente que puede haber más de un cromosoma por nucleoide.
El nucleoide compacta al ADN procariota al menos unas 1000 veces. La estructura topológica misma del nucleoide es parte fundamental de la regulación de los genes que porta el cromosoma. Es decir, estructura y función constituyen una misma unidad.
El ADN en el núcleo eucariota no está desnudo. Interacciona con muchas proteínas, las más importantes de las cuales son las histonas. Las histonas son proteínas pequeñas cargadas positivamente que se unen al ADN de manera no específica.
En el núcleo lo que observamos es un complejo ADN:histonas, al cual llamamos cromatina. La cromatina altamente condensada, que generalmente no se expresa, es la heterocromatina. Por el contrario, la menos compactada (más laxa), o eucromatina, es la cromatina con genes que se expresan.
La cromatina tiene varios niveles de compactación. El más elemental es el del nucleosoma; le siguen la fibra del solenoide y los lazos de cromatina interfásicos. Solo cuando un cromosoma se divide es que se muestran los niveles de compactación máximos.
El nucleosoma es la unidad básica de organización de la cromatina. Cada nucleosoma está formado por un octámero de histonas que forman una especie de tambor.
El octámero está formado por dos copias de cada una de las histonas H2A, H2B, H3 y H4. Alrededor de ellos, el ADN da casi 1.7 vueltas. Le sigue una fracción de ADN libre llamado linker de 20 pb asociado a la histona H1, y luego otro nucleosoma. La cantidad de ADN en un nucleosoma y el que lo une a otro es de unas 166 pares de bases.
Este paso de empaquetamiento del ADN compacta a la molécula unas 7 veces. Es decir, pasamos de un metro a poco más de 14 cm de ADN.
Este empaquetamiento es posible porque las histonas positivas anulan la carga negativa del ADN, y la consecuente autorepulsión electrostática. La otra razón es que el ADN se puede doblar de tal manera que puede darle vueltas al octámero de histonas.
La fibra de cuentas en un collar que forman muchos nucleosomas sucesivos se enrolla adicionalmente en una estructura más compactada.
Aunque no tenemos claro qué estructura realmente adopta, sí sabemos que alcanza un grosor de unos 30 nm. Esta es la denominada fibra de 30 nm; para su formación y estabilidad es fundamental la histona H1.
La fibra de 30 nm es la unidad estructural básica de la heterocromatina. La de los nucleosomas laxos, la de la eucromatina.
La fibra de 30 nm, sin embargo, no es completamente lineal. Por el contrario, forma lazos de unos 300 nm de longitud, de manera serpenteante, sobre una matriz proteica poco conocida.
Estos lazos sobre una matriz proteica forman una fibra de cromatina más compacta de 250 nm de diámetro. Finalmente, se alinean a manera de una hélice simple de 700 nm de grosor dando origen a una de las cromátidas hermanas de un cromosoma mitótico.
Al final, el ADN en la cromatina nuclear se compacta unas 10000 veces en el cromosoma de la célula en división. En el núcleo interfásico su compactación también es alta ya que es de unas 1000 veces comparado con el ADN “lineal”.
En el mundo de la biología del desarrollo se dice que la gametogénesis resetea al epigenoma. Es decir, que borra las marcas del ADN que la vida de quien dio origen al gameto produjo o experimentó.
Estas marcas incluyen la metilación del ADN y las modificaciones covalentes de las histonas (Código de las histonas). Pero no todo el epigenoma se resetea. Lo que queda con marcas será responsable de la impronta genética paterna o materna.
El reseteo implícito a la gametogénesis es más sencillo verlo en los espermatozoides. En los espermatozoides, el ADN no está empaquetado con histonas. Por lo tanto, la información asociada con sus modificaciones en el organismo productor, generalmente, no se heredan.
En los espermatozoides el ADN se empaqueta gracias a la interacción con unas proteínas de unión no específica al ADN llamadas protaminas. Estas proteínas forman puentes disulfuro entre sí, contribuyendo así a formar capas de ADN sobrepuesto que no se repelen electrostáticamente.
- Alberts, B., Johnson, A. D., Lewis, J., Morgan, D., Raff, M., Roberts, K., Walter, P. (2014) Molecular Biology of the Cell (6th Edition). W. W. Norton & Company, New York, NY, USA.
- Annunziato, A. (2008) DNA Packaging: Nucleosomes and chromatin. Nature Education 1:26. (https://www.nature.com/scitable/topicpage/dna-packaging-nucleosomes-and-chromatin-310).
- Brooker, R. J. (2017). Genetics: Analysis and Principles. McGraw-Hill Higher Education, New York, NY, USA.
- Martínez-Antonio, A. Medina-Rivera, A., Collado-Vides, J. (2009) Structural and functional map of a bacterial nucleoid. Genome Biology, doi: 10.1186/gb-2009-10-12-247.
- Mathew-Fenn, R. S, Das, R., Harbury, P. A. B. (2008) Remeasuring the double helix. Science, 17:446-449.
- Travers, A. A. (2004) The structural basis of DNA flexibility. Philosophical Transactions of the Royal Society of London, Series A, 362:1423-1438.
- Travers, A., Muskhelishvili, G. (2015) DNA structure and function. FEBS Journal, 282:2279-2295.
