Operón: descubrimiento, modelo, clasificación, ejemplos
Un operón consiste en un grupo de genes ordenados consecutivamente que se regulan entre sí, que codifican proteínas que están relacionadas funcionalmente y los cuales se encuentran a lo largo del genoma de las bacterias y de los genomas “ancestrales”.
Este mecanismo de regulación fue descrito por F. Jacob y J. Monod en 1961, hecho que les valió el premio nobel de Fisiología y Medicina en 1965. Estos investigadores propusieron y demostraron el funcionamiento de los operones a través de los genes que codifican para las enzimas requeridas por Escherichia coli para la utilización de la lactosa.

Los operones se encargan coordinar la síntesis de proteínas de acuerdo con las necesidades de cada célula, es decir, que solo se expresan para generar unas proteínas en el momento y en el lugar exacto donde son requeridas.
Los genes contenidos dentro de los operones son, por lo general, genes estructurales, lo que quiere decir que codifican para enzimas importantes involucradas de manera directa en las vías metabólicas dentro de la célula. Estas pueden ser la síntesis de aminoácidos, de energía en forma de ATP, de carbohidratos, etc.
Los operones también suelen encontrarse en los organismos eucariotas, sin embargo, en contraste con los organismos procariotas, en los eucariotas la región del operón no se transcribe como una sola molécula de ARN mensajero.
Índice del artículo
El primer avance importante en cuanto a los operones realizado por François Jacob y Jacques Monod fue enfocar el problema de la “adaptación enzimática”, que consistía en la aparición de una enzima específica solo cuando la célula se encontraba en presencia de un sustrato.
Tal respuesta de las células a los sustratos se había observado en las bacterias desde hacía muchos años atrás. Sin embargo, los investigadores se preguntaban cómo la célula determinaba exactamente qué enzima debía sintetizar para metabolizar dicho sustrato.
Jacob y Monod observaron que las células bacterianas, en presencia de carbohidratos de tipo galactosa, producían 100 veces más β-galactosidasa que en condiciones normales. Esta enzima es la que se encarga de descomponer los β–galactósidos para que la célula los aproveche metabólicamente.
Así pues, ambos investigadores llamaron a los carbohidratos de tipo galactósidos como “inductores”, ya que los mismos se encargaban de inducir un incremento en la síntesis de β-galactosidasa.
Así mismo, Jacob y Monod encontraron una región genética con tres genes que se controlaban de forma coordinada: el gen Z, codificante para la enzima β-galactosidasa; el gen Y, codificante para la enzima lactosa permeasa (transporte de los galactósidos); y el gen A, el cual codifica para la enzima transacetilasa, que también es esencial para la asimilación de los galactósidos.
Mediante análisis genéticos posteriores Jacob y Monod aclararon todos los aspectos del control genético del operón lactosa, concluyendo que el segmento de genes Z, Y y A constituye una sola unidad genética con expresión coordinada, que fue lo que definieron como “operón”.
El modelo del operón fue descrito con precisión por primera vez en 1965 por Jacob y Monod para explicar la regulación de los genes que se transcriben y traducen para las enzimas que se requieren en Escherichia coli con el fin de metabolizar a la lactosa como fuente de energía.
Estos investigadores propusieron que las transcripciones del gen o del conjunto de genes que se ubican de forma consecutiva se regulan por dos elementos: 1) un gen regulador o gen represor 2) y un gen operador o secuencia operadora.
El gen operador siempre se encuentra al lado del o los genes estructurales cuya expresión se encarga de regular, mientras que el gen represor codifica para una proteína llamada “represor” que se une al operador e impide su transcripción.
La transcripción es reprimida cuando el represor está unido al gen operador. De este modo la expresión genética de los genes que codifican a las enzimas necesarias para asimilar la lactosa no son expresadas y, por lo tanto, no puede metabolizar dicho disacárido.
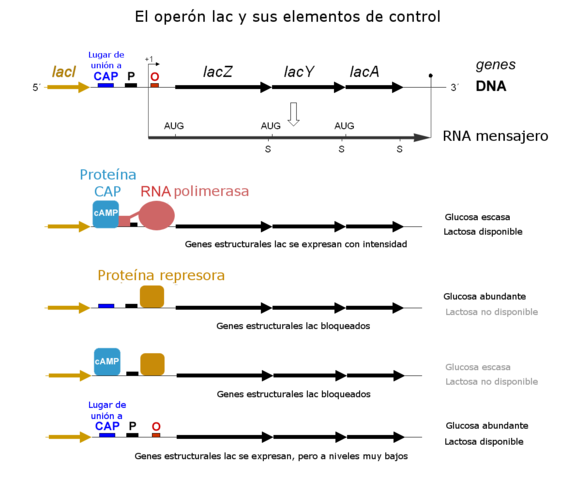
En la actualidad se sabe que la unión del represor al operador evita, con mecanismos estéricos, que la ARN polimerasa se una al sitio promotor para que comience a transcribir los genes.
El sitio del promotor es el “sitio” que reconoce la ARN polimerasa para unirse y transcribir los genes. Al no poder unirse no puede transcribir ninguno de los genes de la secuencia.
El gen operador se encuentra entre una región genética de la secuencia conocida como promotor y los genes estructurales. Sin embargo, Jacob y Monod no identificaron esta región en su época.
Actualmente se sabe que la secuencia completa que incluye al gen o genes estructurales, al operador y al promotor, es en esencia lo que constituye a un “operón”.
Los operones se clasifican solo en tres categorías diferentes que dependen de la forma en que se regulan, es decir, algunos se expresan de forma continua (constitutivo), otros necesitan alguna molécula o factor específico para activarse (inducibles) y otros se expresan continuamente hasta que el inductor se expresa (reprimible).
Los tres tipos de operones son:
Los operones de este tipo se regulan por moléculas en el ambiente como aminoácidos, azúcares, metabolitos, etc. Estas moléculas son conocidas como inductores. Si no se encuentra la molécula que actúa como inductor los genes del operón no se transcriben activamente.
En los operones inducibles, el represor libre se une al operador y evita la transcripción de los genes que se encuentran en el operón. Cuando se une el inductor al represor, se forma un complejo que no puede unirse al represor y por tanto se traducen los genes del operón.
Estos operones dependen de unas moléculas específicas: aminoácidos, azúcares, cofactores o factores de transcripción, entre otras. Estas son conocidas como correpresores y actúan de forma totalmente opuesta a los inductores.
Solo cuando el correpresor se une con el represor, la transcripción se detiene y así la transcripción de los genes contenidos en el interior en el operón no ocurre. Entonces la transcripción de un operón reprimible solo se detiene con la presencia del correpresor.
Este tipo de operones no se regulan. Se transcriben constantemente de forma activa y, en caso de que alguna mutación que afecte la secuencia de estos genes, la vida de las células que los contienen puede verse afectada y, por lo general, desencadenar la muerte celular programada.
El primer y más reconocido ejemplo de la función de un operón es el Operón lac (lactosa). Este sistema se encarga de transformar a la lactosa, un disacárido, en los monosacáridos glucosa y galactosa. En este proceso actúan tres enzimas:
– La β-galactosidasa, encargada de convertir la lactosa en glucosa y galactosa.
– La lactosa permeasa, encargada de transportar la lactosa desde el medio extracelular hacia el interior de la célula y
– La transcetilasa, que pertenece al sistema, pero posee una función desconocida
El operón trp (triptófano) de Escherichia coli controla la síntesis de triptófano, teniendo como precursor al ácido corísmico. Dentro de este operón se encuentran los genes para cinco proteínas que se utilizan para la producción de tres enzimas:
– La primera enzima, codificada por los genes E y D, cataliza las dos primeras reacciones de la vía del triptófano y se conoce como antranilato sintetasa
– La segunda enzima es la glicerolfosfato y cataliza los pasos posteriores a la antranilato sintetasa
– La tercera y última enzima es la triptófano sintetasa, encargada de producir el triptófano a partir de indol-glicerol fosfato y serina (esta enzima es producto de los genes B y A)
- Blumenthal, T. (2004). Operons in eukaryotes. Briefings in Functional Genomics, 3(3), 199-211.
- Gardner, E. J., Simmons, M. J., Snustad, P. D., & Santana Calderón, A. (2000). Principles of genetics. Principios de genética.
- Osbourn, A. E., & Field, B. (2009). Operons. Cellular and molecular life sciences, 66(23), 3755-3775.
- Shapiro, J., Machattie, L., Eron, L., Ihler, G., Ippen, K., & Beckwith, J. (1969). Isolation of pure lac operon DNA. Nature, 224(5221), 768-774.
- Suzuki, D. T., & Griffiths, A. J. (1976). An introduction to genetic analysis. WH Freeman and Company.
